RでStan(事後予測チェック)
外部ライブラリの準備
install.packages("bayesplot") install.packages("dplyr") library(bayesplot)
MCMCの結果のプロットに便利なbayesplotを入れます.なぜか依存パッケージのdplyrが自動で入らなかったので別途入れる必要がありました.
問題設定
ある小動物の発見個体数のモデル化に取り組みます.
群れを作ることなくランダムに生息している動物がある観測地点において発見された個体数を扱います.
個の場合,小動物の発見個体数データが得られる確率的な家庭はポアソン分布だと考えられます.
そこで,ポアソン分布でモデル化するケースと,あえて誤ったモデルとして正規分布でモデル化するケースを考えます.
実装
Rファイル
library(rstudioapi) library(rstan) library(bayesplot) rstan_options(auto_write=TRUE) # 2回目以降のコンパイルを不要にする options(mc.cores=parallel::detectCores()) # 計算の並列化 animal_num <- read.csv("2-5-1-animal-num.csv") data_list <- list(animal_num=animal_num$animal_num, N=nrow(animal_num)) mcmc_normal <- stan( file = "2-5-1-normal-dist.stan", data = data_list, seed = 1 ) mcmc_poisson <- stan( file = "2-5-1-poisson-dist.stan", data = data_list, seed = 1 )
Stanファイル
正規分布
data {
int N;
vector[N] animal_num;
}
parameters {
real<lower=0> mu;
real<lower=0> sigma;
}
model {
animal_num ~ normal(mu, sigma);
}
generated quantities{
// 事後予測分布を得る
vector[N] pred;
for (i in 1:N){
pred[i] = normal_rng(mu, sigma);
}
}
ポアソン分布
data {
int N;
int animal_num[N];
}
parameters {
real<lower=0> lambda; // 強度
}
model {
// 強度lambdaのポアソン分布
animal_num ~ poisson(lambda);
}
generated quantities{
// 事後予測分布を得る
int pred[N];
for (i in 1:N){
pred[i] = poisson_rng(lambda);
}
}generated quantitiesブロックは「モデル推定以外の目的で乱数を得たいモノ」を指定するブロックで,モデルを用いた予測値などをここに記述します.
normal_rng,poisson_rngメソッドを使うことで正規分布,ポアソン分布に従う乱数をpredに格納します.
結果の確認
y_rep_normal <- rstan::extract(mcmc_normal)$pred y_rep_poisson <- rstan::extract(mcmc_poisson)$pred ppc_hist(y=animal_num$animal_num, yrep = y_rep_normal[1:5,]) ppc_hist(y=animal_num$animal_num, yrep = y_rep_poisson[1:5,])

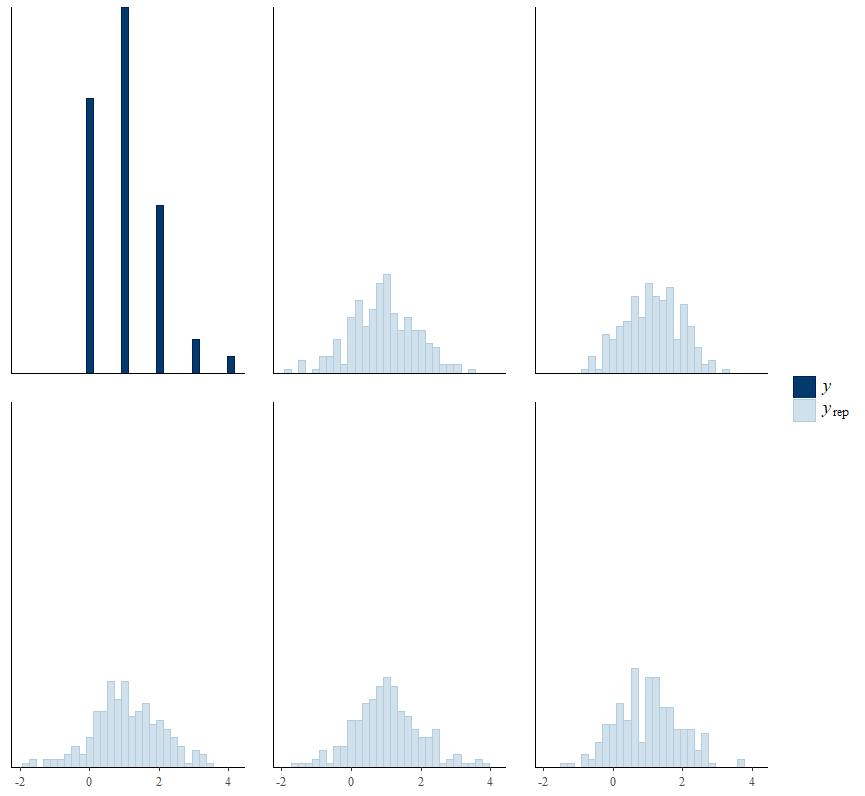
観測データの分布と,1~5回目のMCMCサンプルのヒストグラムを比較することで,ポアソン分布でもモデル化した方はそれっぽい結果になっているのに対し,正規分布でモデル化した方は観測データの分布と大きく異なっていることが確認できます.